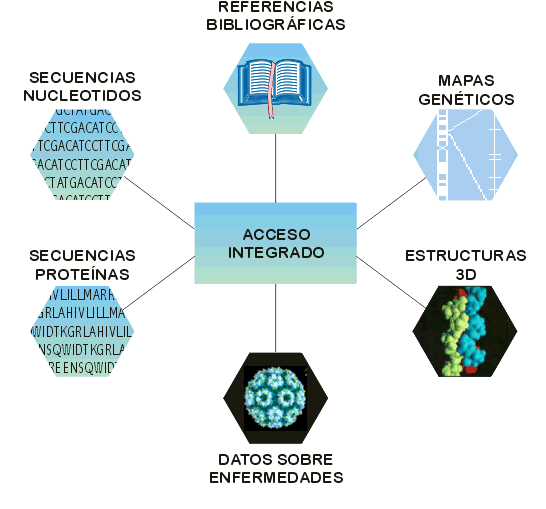
La Bioinformática es un campo interdisciplinario que se encuentra en la intersección entre las Ciencias de la Vida y de la Información, proporcionando herramientas y recursos para favorecer la Investigación Biomédica. La Bioinformática se orienta hacia la investigación y desarrollo de herramientas útiles para llegar a entender el flujo de información desde los genes a las estructuras moleculares, a su función bioquímica, a su conducta biológica y, finalmente, a su influencia en las enfermedades y en la salud.Históricamente, el uso de los ordenadores para resolver cuestiones biológicas comenzó con el desarrollo de algoritmos y su aplicación en el entendimiento de las interacciones de los procesos biológicos y las relaciones entre diversos organismos. El incremento exponencial en la cantidad de secuencias disponibles, así como la complejidad de las técnicas que emplean los ordenadores para la adquisición y análisis de datos, han servido para la expansión de la bioinformática.En los últimos años, la bioinformática ha trabajado con muchas bases de datos que almacenaban información biológica a medida que iba apareciendo. Esto no sólo ha tenido efectos positivos: muchos científicos se quejan de la creciente complejidad que representa encontrar información útil en este "laberinto de datos”. Para mejorar esta situación, se desarrollan técnicas que integran la información dispersa, gestionan bases de datos distribuidas, las seleccionan automáticamente, evalúan su calidad, y facilitan su accesibilidad para los investigadores. Se habla de Bioinformática Integradora. En ella no deben faltar ayudas para la navegación por la información, que cada vez, con más énfasis, reside en Internet y no en bases de datos locales.
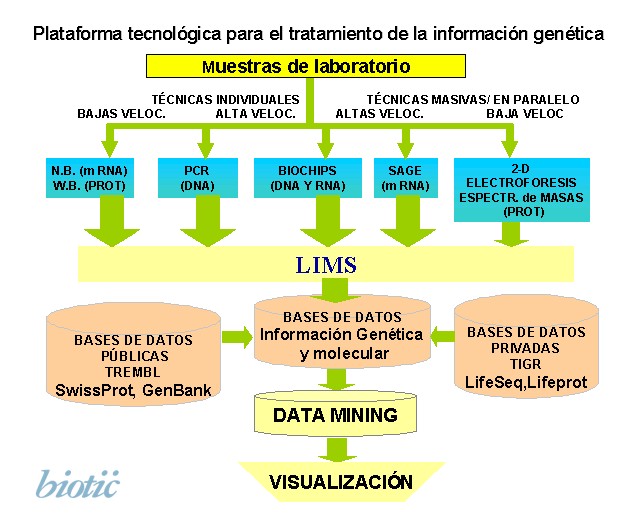
Los procesos celulares son gobernados por el repertorio de genes expresados y su patrón de actividad temporal. Se necesitan herramientas para gestionar información genética en paralelo. Para ello se emplean nuevas tecnologías para extracción de conocimiento, minería de datos y visualización. Se aplican técnicas de descubrimiento de conocimiento a problemas biológicos como análisis de datos del Genoma y Proteoma. La bioinformática, en este sentido, ofrece la capacidad de comparar y relacionar la información genética con una finalidad deductiva, siendo capaz de ofrecer unas respuestas que no parecen obvias a la vista de los resultados de los experimentos . Todas estas tecnologías vienen justificadas por la necesidad de tratar información masiva, no individual, sino desde enfoques celulares integrados (genómica funcional, proteómica, expresión multigénica,...). Los sistemas LIMS permiten la integración y gestión de los datos de laboratorio.Nuevas tecnologías para el tratamiento de la información genética
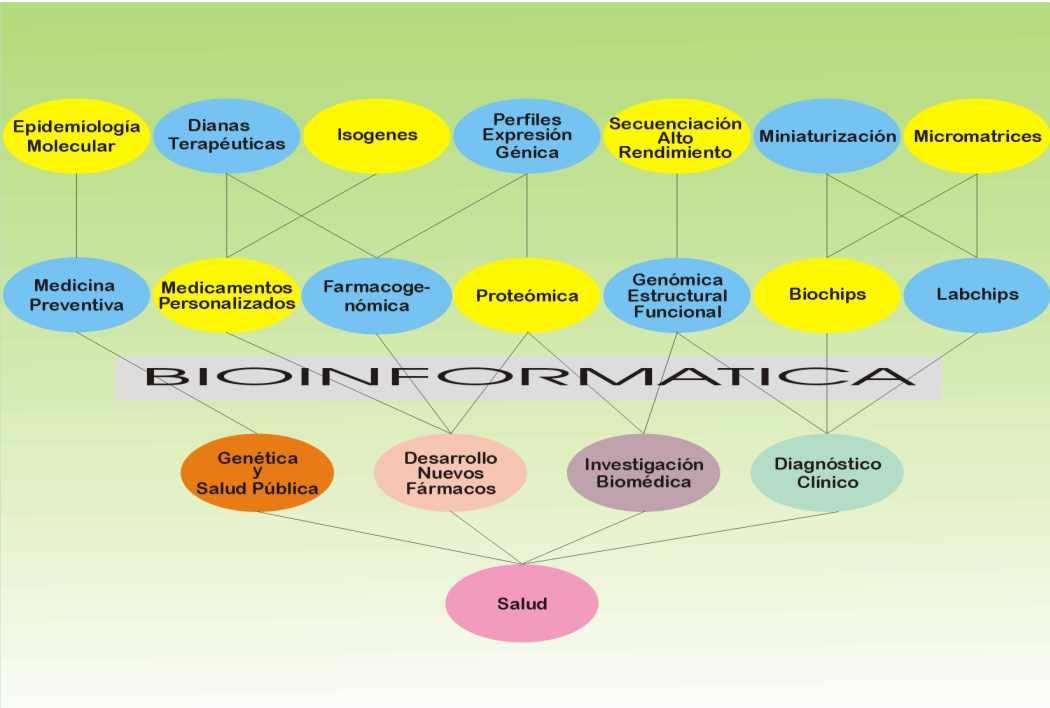
La explotación de la información genómica individual va a posibilitar nuevas técnicas útiles para la investigación de enfermedades y el diagnóstico clínico, esta faceta representa otro carácter diferencial de la nueva Bioinformática, su clara orientación hacia la resolución de problemas de Salud, de donde recibe el adjetivo de Bioinformática Aplicada.
Áreas de interés para la nueva bioinformática aplicada a la Salud
Impacto en la Investigación Biomédica
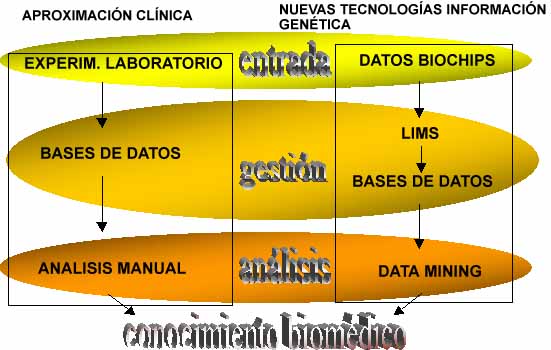
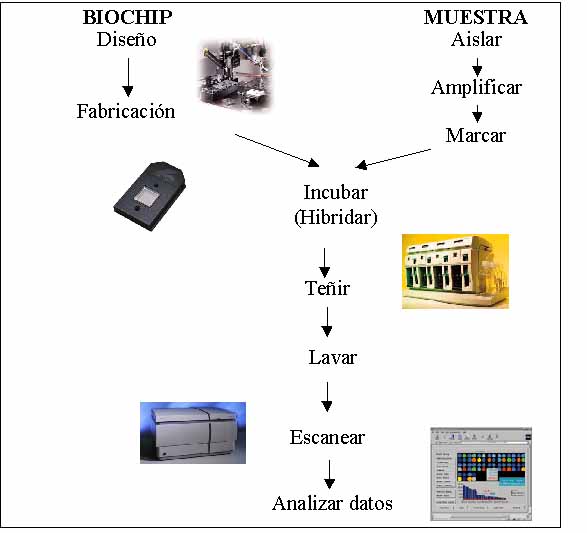
La aplicación de las nuevas tecnologías de la información genética en el entorno de investigación biomédica ha dado lugar a la aparición de un nuevo vocablo: "Biología in silico": obtención de conocimiento mediante consideraciones teóricas, simulaciones y experimentos llevados a cabo sobre la tecnología basada en silicio de un ordenador. En la figura de abajo se aprecia como las nuevas técnicas permiten que todo el flujo de trabajo de la experimentación biológica sea llevado a cabo de un modo asistido por ordenador.
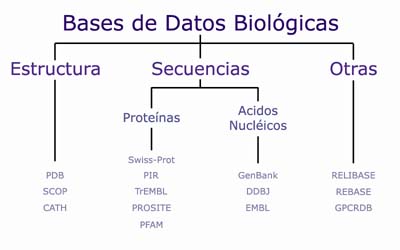
Base de Datos BiológicaUna base de datos biológica se puede definir como una colección de datos crudos, teóricos o experimetnales que está organizada y almacenada en sitios electrónicos. Los contenidos de dicha información deberán ser fácilmente recuperables, manejables, accesibles y renovables. Las bases de datos biológicas se pueden clasificar de manera amplia cómo:
Proyecto Genoma Humano (HGP)La palabra GENOMA fue usada por primera vez por H. Winkler en 1920, y está formada por la elisión de las palabras GEN y cromosOMA. Eso es justamente lo que significa: el conjunto completo de los cromosomas (y los genes que estos portan) de un organismo.
El objetivo del Proyecto Genoma Humano es mapear y secuenciar los 60.000 - 100.000 genes de que consta el patrimonio genético de la especie humana.
La propuesta para secuenciar el genoma humano surgió en 1985/86. Siguió una intensa discusión que desembocó en 1988 en la propuesta formal de un plan a 15 años, recomendando la estrategia de 'primero el mapa, después la secuencia'.



Cadena de ARN
Algoritmos de Decodificación de Secuencias
BLAST es el algoritmo utilizado por una familia de cinco programas que alinean una secuencia desconocida contra las almacenadas en una base de datos molecular. Con la finalidad de evaluar la sigificancia del las comparaciones se utilizan métodos estadísticos. Los alineamientos que son reportados (por Ej.. aquellas secuencias en la base de datos que son parecidas o iguales a la desconocida) se organizan en orden de significancia de mayor a menor. El algoritmo BLAST ha sido optimizado para alineamientos de secuencias pero no para la búsqueda de motivos.
Naves Microcóspicas
Investigadores financiados por una donación de la NASA, comenzaron recientemente un proyecto para hacer realidad estos escenarios futuristas. Si tienen éxito, las "naves" desarrolladas por los científicos -- llamadas nanopartículas o nanocápsulas -- podrían ayudar a hacer realidad otra historia de ciencia ficción: la exploración humana de Marte y la permanencia a largo plazo en el espacio por los humanos.
Ni siquiera los materiales más avanzados, utilizados para protegerse de la radiación en las naves espaciales, son capaces de aislar completamente a los astronautas de la radiación de alta energía del espacio. Estos fotones y partículas atraviesan sus cuerpos como balas infinitesimales, destruyendo moléculas a su paso. Cuando el ADN sufre daños por esta radiación, las células se comportan erráticamente, ocasionalmente generando cánceres.
Si el daño causado por la radiación es muy grande, las nanopartículas podrían entrar en las células dañadas y liberar enzimas que inicien la "secuencia auto-destructiva" de la célula, llamada apoptosis. Si el daño no es muy extenso, pueden soltar enzimas reparadoras de ADN para intentar arreglar la célula y hacer que vuelva a funcionar normalmente.
Minúsculas cápsulas, mucho más pequeñas que estas células de sangre, podrían ser un día inyectadas en la corriente sanguínea de las personas, para tratar enfermedades que van desde el cáncer a daños por radiación. Derechos Reservados 1999, Daniel Higgins, Universidad de Illinois en Chicago. La radiación cósmica de alta energía puede producir daños en el ADN y hacer que las células se comporten erráticamente. Imagen cortesía de NASA/OBPR.
Una membrana de dos capas separa el interior de la célula, abajo a la derecha de esta imagen, del ambiente que la rodea. Las moléculas complejas en esta membrana exterior controlan la forma como el interior de la célula interactúa con su ambiente. Imagen, derechos reservados, Scott Barrows, Universidad de Illinois en Chicago.
Proyecto INFOBIOCHIP
INFOBIOCHIP se construye gracias a la enorme cantidad de información recopilada, filtrada y organizada dentro de este campo tecnológico desde esa fecha. El volcado de toda esta información en INTERNET permite una fácil y veloz actualización de la misma en respuesta a la rapidísima evolución de este sector tecnológico.
INFOBIOCHIP surge como consecuencia del interés y continuo seguimiento que de estas tecnologías ha mantenido la Unidad de Bioinformática del Instituto de Salud Carlos III desde 1997.
A finales de los años 80, la tecnología que desembocaría en la plataforma GeneChip fue desarrollada por cuatro científicos, en Affymax: Stephen Fodor, Michael Pirrung, Leighton Read y Lubert Stryer. El proyecto original estaba destinado a la construcción de péptidos sobre chips, pero desembocó en la capacidad para construir secuencias de DNA sobre chips. La aplicación práctica de esta idea se llevó a cabo por la empresa Affymetrix, que comenzó a actuar como una compañía independiente en el año 1993.